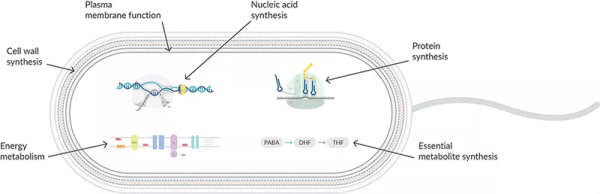
Proteinsynthese-Inhibitoren wirken am Ribosom zu verschiedenen Zeitpunkten der prokaryotischen Translation (Abbildung 1). Sie sind hochselektiv und wirken nur auf 70S-Ribosomen in prokaryotischen Zellen. Denn Eukaryotische Zellen weisen eine andere ribosomale Größe, Sequenz, Struktur und ein anderes Verhältnis von Protein zu RNA auf. Bei der Translation in Prokaryoten werden eine kleine Untereinheit (30S) und eine große Untereinheit (50S) zu einem Ribosom zusammengesetzt, das die mRNA bindet, um die Nukleotidsequenz zu lesen. tRNAs binden an die A-, P- und E-Stellen während der Initiations-, Elongations- und Terminationsphasen, um die Polypeptidsequenz zu translatieren. Indem man auf verschiedene Phasen der mRNA-Translation abzielt, können Antibiotika ausgetauscht werden, wenn sich eine Resistenz entwickelt.

Abbildung 1: Antibiotika greifen in verschiedene Phasen der Proteinsynthese ein.
Tetracycline binden reversibel an die 30S ribosomale Untereinheit an einer Position, die die Bindung der tRNA an die Akzeptorstelle am mRNA-Ribosomenkomplex blockiert. Die Resistenz gegen Tetracycline entwickelt sich in erster Linie durch die Verhinderung der Akkumulation des Wirkstoffs in der Zelle. Dies geschieht entweder durch Verringerung des Influx oder durch Erhöhung des Efflux.
Aminoglykoside und Aminonukleoside wie Puromycin binden an das aktive Zentrum der 16S rRNA innerhalb der 30S ribosomalen Untereinheit und stören die genaue Erkennung der tRNA durch die rRNA während der Translation. Die anfänglichen Schritte, die für die Peptidsynthese erforderlich sind, wie z. B. die Bindung der mRNA und die Assoziation der 50S ribosomalen Untereinheit, werden nicht unterbrochen. Die fehlerfreie Elongation scheitert jedoch aufgrund der beeinträchtigten Translationsgenauigkeit, was zu einer vorzeitigen Terminierung der naszierenden Polypeptidketten führt. Im Falle von Puromycin wurden diese Eigenschaften in Assays zur Peptidbindungsbildung und Elongation sowie als Selektionsmittel in Zellkultursystemen ausgenutzt. Die Resistenz gegen Aminoglykoside entwickelt sich aus strukturellen Modifikationen des Antibiotikums durch Aminoglykosid-Phosphotransferasen, Aminoglykosid-Nukleotidyltransferasen und Aminoglykosid-Acetyltransferasen, die in resistenten Organismen exprimiert werden.
Oxazolidinone binden an der P-Stelle der ribosomalen 50S-Untereinheit. Dort hemmen sie die Bindung der tRNA und verhindern die Bildung des Initiationskomplexes. Wenn der 70S-Komplex bereits gebildet ist, hemmt die Bindung von Oxazolidinonen die Translokation der Peptidkette von der A-Stelle zur P-Stelle während der Bildung der Peptidbindung. Mutationen an der 23S rRNA innerhalb der 50S ribosomalen Untereinheit können zu einer reduzierten Affinität für Oxazolidinone führen. So kann eine Resistenz verursacht werden.
Amphenicole blockieren den Peptidyl-Transfer-Schritt der Elongation an der 50S-Untereinheit von Bakterien, wobei sie höchstwahrscheinlich an der A-Stelle wirken. Sie können auch zusätzliche ribosomale Funktionen stören, wie z. B. die Termination, die Translationsgenauigkeit und die Synthese der 50S ribosomalen Untereinheiten. Chloramphenicol kann auch an das mitochondriale 70S-Ribosom binden. Dadurch wird die mitochondriale Proteinsynthese gehemmt, wodurch Toxizität in Eukaryoten entsteht. Mutationen oder Veränderungen am Target von Amphenicolen sind ein häufiger Mechanismus der Resistenz.
Lincosamide wirken wie strukturelle Analoga von Teilen der tRNA vor der Translokation in der Elongationsphase und unterbrechen den Prozess der Initiierung der Peptidkette in der 50S-Untereinheit. Sie können auch die Dissoziation der Peptidyl-tRNA von den Ribosomen bewirken. Die Resistenz gegen Lincosamide kann durch Methylierung der 23S rRNA, Modifikation der Antibiotika durch spezifische Enzyme oder aktive Effluxsysteme verursacht werden.
Die meisten Macrolide interagieren mit der 23S rRNA innerhalb der 50S ribosomalen Untereinheit und blockieren den Zugang zum Ausgangstunnel des elongierenden Peptids. Dies führt zu einer vorzeitigen Freisetzung von Peptidyl-tRNA-Intermediaten. Sie konkurrieren mit Lincosamid-Antibiotika, die direkte Peptidyltransferase-Inhibitoren sind. Zu den drei Hauptmechanismen, die für die erworbene Makrolidresistenz verantwortlich sind, gehören die Modifiktation des Ribosoms, ein aktiver Efflux-Mechanismus und eine Mutation in der 23S rRNA.
Streptogramine wirken je nach Typ ähnlich wie Lincosamide und Makrolide. Streptogramine vom Typ A blockieren die Bindung des Substrats an die Donor- und Akzeptorstellen des Peptidyltransferasezentrums auf der 50S-Untereinheit und verhindern so früh die Elongation. Streptogramine vom Typ B konkurrieren mit Makroliden um die Bindung an das 50S-Ribosom, um die Bildung langer Polypeptide zu stören und eine vorzeitige Abspaltung unvollständiger Peptidketten zu bewirken. Streptogramine vom Typ A können die Ribosomenaffinität für Streptogramine vom Typ B erhöhen, wodurch eine bakterizide Synergie zwischen den beiden normalerweise bakteriostatischen Wirkstoffen entsteht. Die Resistenz gegen diese Antibiotika beruht hauptsächlich auf der Inaktivierung von Inhibitoren und Mutationen von ribosomalen Proteinen oder rRNA-Genen.
Nitrofurane haben einen einzigartigen Mechanismus zur Verlangsamung des bakteriellen Wachstums. Sie sind Prodrugs, die eine reduktive Aktivierung durch zwei redundante bakterielle Flavoproteine zu reaktiven Intermediaten erfordern. Diese reaktiven Zwischenprodukte haben verschiedene Wirkungen: Sie lösen DNA-Läsionen aus, induzieren oxidativen Stress und hemmen die RNA- und Proteinsynthese. Es ist nicht bekannt, ob die reaktiven Zwischenprodukte DNA, RNA und Proteine direkt modifizieren oder ob sie in erster Linie die zellulären Bestandteile angreifen, die für die Replikation, Transkription, Translation und den Energiestoffwechsel verantwortlich sind. Selten können Mutationen in Genen, die für die Prodrug-aktivierenden Enzyme kodieren, zu Resistenzen führen.